Sintesi dell’articolo “MSH3 Homology and Potential Recombination Link to SARS-CoV-2 Furin Cleavage Site”, pubblicato su Frontiersin.org
Tra le numerose differenze di mutazione puntiforme tra il coronavirus SARS-CoV-2 e il pipistrello RaTG13, solo il sito di scissione della furina a 12 nucleotidi (FCS) supera i 3 nucleotidi. Una ricerca BLAST ha rivelato che una porzione di 19 nucleotidi del genoma SARS.Cov2 che comprende il sito di scissione furina è una corrispondenza complementare al 100% a una sequenza proprietaria ottimizzata per i codoni che è il complemento inverso dell’omologo mutS umano (MSH3). La sequenza inversa del complemento presente in SARS-CoV-2 può verificarsi in modo casuale ma devono essere considerate altre possibilità. La ricombinazione in un ospite intermedio è una spiegazione improbabile. I virus a RNA a filamento singolo come SARS-CoV-2 utilizzano modelli di RNA a filamento negativo nelle cellule infette, che potrebbero portare attraverso la ricombinazione della scelta della copia con un RNA SARS-CoV-2 di senso negativo all’integrazione del filamento negativo MSH3, compreso l’FCS, nel genoma virale. In ogni caso, la presenza della sequenza di RNA lungo 19 nucleotidi che include l’FCS con il 100% di identità al complemento inverso dell’mRNA di MSH3 è altamente insolita e richiede ulteriori indagini.
Introduzione
Sulla base di una recente pubblicazione che descrive le varianti di inserimento di SARS-CoV-2 (1), vorremmo portare l’attenzione sui nostri recenti risultati relativi alla sequenza del sito di scissione della furina (FCS) in SARS-CoV-2 Spike (S) proteina. Il SARS-CoV-2 che causa la pandemia di COVID-19 (2) ha l’82,3% di identità di aminoacidi per combattere il coronavirus SL-CoVZC45, il 77,2% di identità di amminoacidi per SARS-CoV e il 96,2% di identità della sequenza del genoma per combattere il coronavirus RaTG13. Sebbene esistano numerose differenze di mutazione puntiforme tra SARS-CoV-2 e RaTG13, solo un’inserzione e una dissomiglianza superiore a 3 nucleotidi (nt): un’inserzione di 12 nucleotidi che codifica per quattro amminoacidi (aa 681-684, PRRA) nel SARS-CoV È stata scoperta la proteina -2 S. Questo FCS polibasico differenzia SARS-CoV-2 da altri betacoronavirus di linea b o da qualsiasi altro sarbecovirus (3). Un’aggiunta FCS ha migliorato l’infettività di SARS Co-V-2 nel 2019 (4). L’assenza di questo FCS si traduce in varianti SARS-CoV-2 attenuate utili per la vaccinazione animale, accentuandone la rilevanza per l’infezione umana (5). Questo FCS è vitale per la trasmissione umana e del furetto (6), espande il trofismo virale alle cellule umane (7) ed è necessario per malattie gravi in due modelli animali di SARS-CoV-2 (8).
SARS-CoV-2 Spike Protein e MSH3
Una caratteristica peculiare della sequenza nucleotidica che codifica per il sito di scissione della furina PRRA nella proteina SARS-CoV-2 S sono i suoi due codoni CGG consecutivi. Questo codone di arginina è raro nei coronavirus: l’utilizzo del codone sinonimo relativo (RSCU) di CGG nel pangolino CoV è 0, nel pipistrello CoV 0,08, nel SARS-CoV 0,19, nel MERS-CoV 0,25 e nel SARS-CoV-2 0,299 (9).
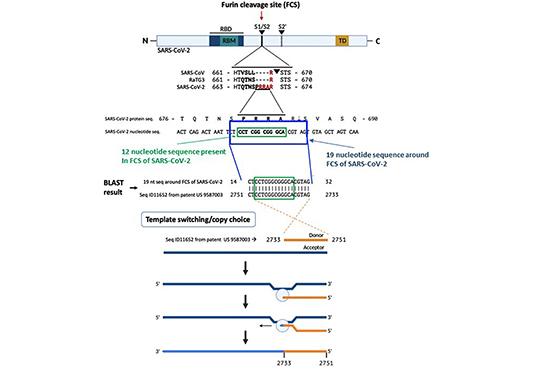
Una ricerca BLAST per l’inserimento di 12 nucleotidi ci ha portato a una corrispondenza inversa del 100% in una sequenza proprietaria (SEQ ID11652, nt 2751-2733) trovata nel brevetto statunitense 9.587.003 depositato il 4 febbraio 2016 (10) (Figura 1) . L’esame di SEQ ID11652 ha rivelato che la corrispondenza si estende oltre l’inserimento di 12 nucleotidi a una sequenza di 19 nucleotidi: 5′-CTACGTGCCCGCCGAGGAG-3′ (nt 2733-2751 di SEQ ID11652), in modo tale che l’mRNA risultante avrebbe 3′-GAUGCACGGGCGGCUCCUC -5′, o equivalentemente 5′- CU CCU CGG CGG GCA CGU AG-3′ (nucleotidi 23547-23565 nel genoma SARS-CoV-2, in cui i quattro codoni in grassetto producono PRRA, amminoacidi 681–684 del suo picco proteina). Questo è molto raro nel database NCBI BLAST.
La correlazione tra questa sequenza SARS-CoV-2 e il complemento inverso di una sequenza proprietaria di mRNA è di origine incerta. L’analisi biostatistica convenzionale indica che la probabilità che questa sequenza sia presente casualmente in un genoma virale di 30.000 nucleotidi è 3,21 × 10 -11 (Figura 2).
La sequenza proprietaria SEQ ID11652, letta nella direzione in avanti, codifica per una corrispondenza di amminoacidi al 100% con l’omologo 3 di mutazione umana S (MSH3) (9). MSH3 è una proteina di riparazione del disadattamento del DNA (parte del complesso MutS beta) (11). SEQ ID11652 viene trascritto in un mRNA MSH3 che sembra essere un codone ottimizzato per l’uomo (12). Non abbiamo trovato la sequenza di 19 nucleotidi CTCCTCGGCGGGCACGTAG in nessun genoma eucariotico o virale ad eccezione di SARS-CoV-2 con copertura e identità del 100% nel database BLAST (tabelle supplementari 1-3).
Discussione
La sostituzione di MSH3 con una sequenza di mRNA ottimizzata per il codone per l’espressione umana ha probabilmente applicazioni nei tumori con carenze di riparazione del disadattamento. Mentre una parte di una sequenza inversa del complemento presente
in SARS-CoV-2 potrebbe essere una coincidenza casuale, altre possibilità meritano di essere prese in considerazione.
È noto che la sovraespressione di MSH3 interferisce con la riparazione del disadattamento (il sequestro di MSH2 dal complesso MutS alfa comprendente MSH2 e MSH6 provoca la degradazione di MSH6 e l’esaurimento di MutS alfa) (13), che ha un’importanza virologica. L’induzione del deficit di riparazione del disadattamento del DNA provoca la permissività dell’infezione da virus dell’influenza A (IAV) delle cellule respiratorie umane e una maggiore patogenicità (14). La carenza di riparazione della mancata corrispondenza può estendere la diffusione di SARS-CoV-2 (15, 16).
L’assenza di CTCCTCGGCGGGCACGTAG da qualsiasi genoma eucariotico o virale nel database BLAST rende la ricombinazione in un ospite intermedio una spiegazione improbabile per la sua presenza in SARS-CoV-2. Un mRNA ottimizzato per il codone umano che codifica per una proteina omologa al 100% all’MSH3 umano potrebbe, nel corso della ricerca virale, indurre inavvertitamente o intenzionalmente un deficit di riparazione del disadattamento in una linea cellulare umana, che aumenterebbe la suscettibilità all’infezione virale simile alla SARS. L’infezione di cellule umane trasdotte con SEQ ID11652-MSH3 da un virus simile alla SARS potrebbe consentire la ricombinazione della scelta della copia (15). La replica di SARS-CoV-2 e di altri virus a RNA a filamento singolo con un genoma di RNA di polarità positiva viene avviata dalla sintesi di RNA a filamento negativo nel citoplasma delle cellule infette (17) (figura 1 ). L’RNA a filamento negativo è un modello per la sintesi di RNA a filamento positivo utilizzato per la traduzione di proteine non strutturali, il complesso di replicazione e trascrizione o nuovi capsidi di virioni. I coronavirus generano RNA a doppio filamento in una fase iniziale dell’infezione attraverso la replicazione genomica e la trascrizione dell’mRNA (18).
L’acquisizione della sequenza FCS del complemento inverso da un mRNA MSH3 di senso positivo sovraespresso potrebbe avvenire attraverso la ricombinazione della scelta della copia con un intermedio di RNA SARS-CoV-2 di senso negativo (15), che comporta il salto da un modello all’altro (19) (Figura 1). L’omologia tra SARS-CoV-2 e altri coronavirus noti è interrotta e la maggior parte delle sequenze SARS-CoV-2 deriva da un antenato comune relativamente recente con il pipistrello RaTG13. Inoltre, i diagrammi di somiglianza (SimPlots) hanno identificato improvvisi cambiamenti nell’identità di sequenza tra SARS-CoV-2 e RaTG13, segnalando potenziali eventi di ricombinazione, che potrebbero spiegare la capacità di SARS-CoV-2 di legarsi ad ACE2 attraverso il suo RBD, che non è il caso per RaTG13 RBD (15).
Una critica a questa ipotesi è che la sequenza identificata si trova sul filo opposto del frame di lettura aperto in SEQ ID11652. Tuttavia, le cellule trasfettate con MSH3, che inducono un deficit di riparazione del disadattamento, potrebbero aver preso di mira il cDNA a doppio filamento che codifica SEQ ID11652. Tali cellule co-trasfettate con un virus simile alla SARS che esprime RdRp potrebbero attaccarsi a questa sequenza di 19 nucleotidi (15) e consentire l’integrazione di un frammento dal filamento negativo nel genoma virale, compreso l’FCS, nonostante si trovino sul filamento opposto della cornice di lettura aperta. I meccanismi di riparazione del disadattamento hanno consentito l’integrazione di brevi frammenti da filamenti antisenso in modelli sperimentali (20, 21). La microomologia può dirigere la ricombinazione tra l’MSH3 e un virus simile alla SARS, che potrebbe aver luogo nella sequenza di 19 nucleotidi di interesse.
La presenza in SARS-CoV-2 di una sequenza di RNA a 19 nucleotidi che codifica per un FCS all’amminoacido 681 della sua proteina spike con il 100% di identità al complemento inverso di una sequenza proprietaria di mRNA MSH3 è altamente insolita. Le potenziali spiegazioni di questa correlazione dovrebbero essere ulteriormente studiate.
Dichiarazione sulla disponibilità dei dati
In questo studio sono stati analizzati set di dati pubblicamente disponibili. Questi dati possono essere trovati qui: SEQ ID11652.